El dolor lumbar no solo es un trastorno sin también un síntoma de varios tipos diferentes de problemas médicos.
Por lo general, resulta de un problema con una o más sectores de la parte inferior de la espalda, como son:
- Ligamentos
- Músculos
- Nervios
- Las estructuras óseas que forman la columna vertebral, llamadas cuerpos vertebrales o vértebras. El dolor de espalda también puede ocurrir debido a un problema con órganos cercanos, como los riñones.
En esta EDA voy a utilizar el dataset de los síntomas del dolor de la parte inferior de la espalda y tratar de descubrir ideas interesantes a partir de este dataset. ¡Comencemos!
Descripción del dataset
El dataset contiene:
-
- 310 Observaciones
- 12 Características
- 1 Etiqueta
Para obtener el dataset pulsa aquí
Importar los paquetes necesarios:
import pandas as pd
import matplotlib.pyplot as plt
%matplotlib inline
import seaborn as sns
sns.set()
from sklearn.preprocessing import MinMaxScaler, StandardScaler, LabelEncoder
from sklearn.svm import SVC
from sklearn.naive_bayes import GaussianNB
from sklearn.linear_model import LogisticRegression
from xgboost import XGBClassifier, plot_importance
from sklearn.model_selection import train_test_split
from sklearn.metrics import accuracy_score,confusion_matrix
Leyendo el archivo .csv:
dataset = pd.read_csv("../input/Dataset_spine.csv")
Viendo las 5 filas superiores del dataset:
dataset.head() # this will return top 5 rows
Eliminando columna dummy:
# This command will remove the last column from our dataset.
del dataset["Unnamed: 13"]
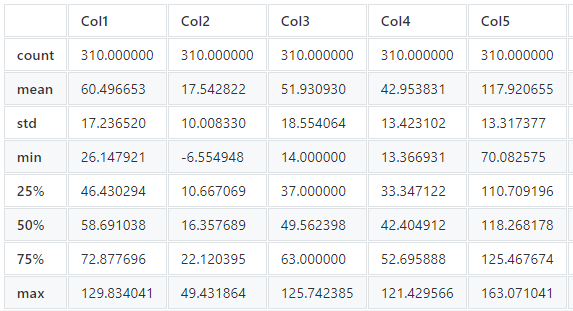
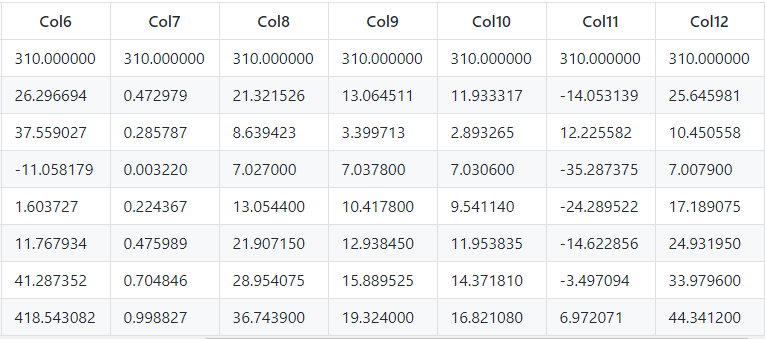
Resumen del dataset:
El método DataFrame.describe () genera estadísticas descriptivas que resumen la tendencia central, la dispersión y la forma de la distribución de un dataset, excluyendo los valores NaN.
Este método nos dice muchas cosas sobre un dataset. Una cosa importante es que el método describe() trata solo con valores numéricos. No funciona con ningún valor categórico.
Ahora, comprendamos las estadísticas que se generan con el método describe():
countnos dice el número de filasNoN-emptyen una función.meannos da el valor medio de esa característica.stddice el Valor de Desviación Estándar de esa característica.minmuestra el valor mínimo de esa característica.25%,50%, y75%son el percentil / cuartil de cada característica. Esta información cuartil nos ayuda a detectar Valores atípicos .- max nos dice el valor máximo de esa función.
dataset.describe()
Para ver el dataset completo pulsa aquí
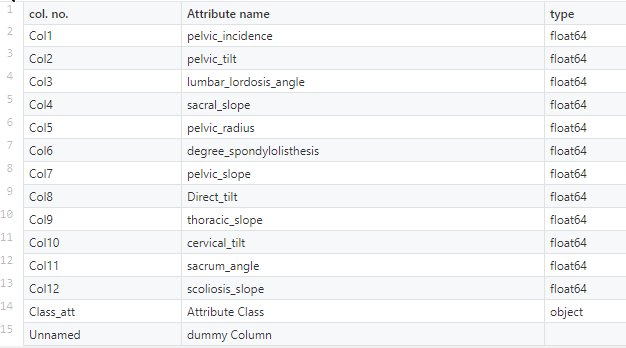
Cambiar el nombre de la columna para aumentar la legibilidad:
dataset.rename(columns = {
"Col1" : "pelvic_incidence",
"Col2" : "pelvic_tilt",
"Col3" : "lumbar_lordosis_angle",
"Col4" : "sacral_slope",
"Col5" : "pelvic_radius",
"Col6" : "degree_spondylolisthesis",
"Col7" : "pelvic_slope",
"Col8" : "direct_tilt",
"Col9" : "thoracic_slope",
"Col10" :"cervical_tilt",
"Col11" : "sacrum_angle",
"Col12" : "scoliosis_slope",
"Class_att" : "class"}, inplace=True)
DataFrame.info () imprime información sobre un DataFrame incluyendo el tipo de index y los dtypes de column , valores non-nulll y uso de la memoria. Podemos usar info() para saber si un dataset contiene algún valor faltante o no.
dataset.info ()
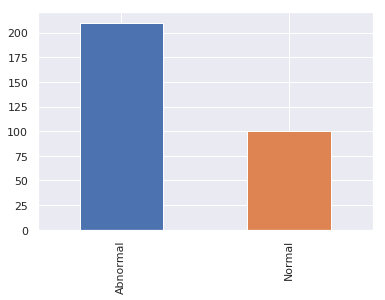
Visualiza el número de casos anormales y normales:
La tendencia de los casos abnormal es 2 veces superior a los casos normal .
dataset ["class"] .value_counts (). sort_index (). plot.bar ()
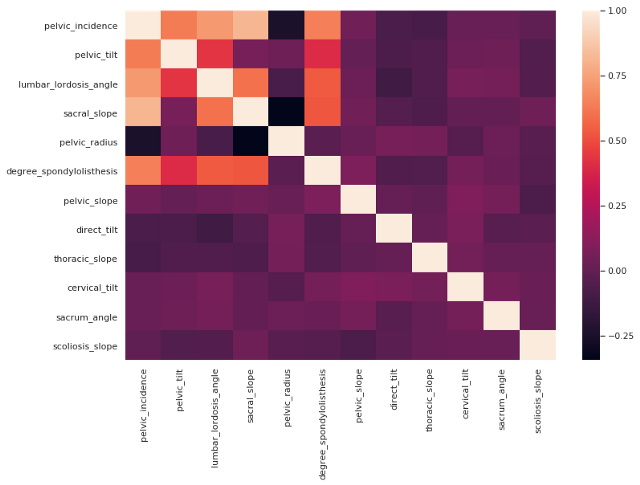
Comprobando la correlación entre features o características:
Coeficiente de correlación es una medida numérica de algún tipo de correlación, es decir, una relación estadística entre dos variables.
dataset.corr ()
Visualiza la correlación con heatmap :
plt.subplots ( figsize = (12,8))
sns.heatmap (dataset.corr ())
Correlograma personalizado:
Un pair plot nos permite ver ambos distribución de variables individuales y relaciones entre dos variables.
sns.pairplot (dataset, hue = "class")
Muchas cosas están sucediendo en el pair plot de abajo. Vamos a tratar de entender el pair plot.
first row X first column, esta diagonal nos muestra la distribución de pelvic_incidence.Del mismo modo, si nos fijamos en lasecond row X second columndiagonal podemos ver la distribución de pelvic_tilt.Todas las celdas, excepto las diagonales, muestran la relación entre una entidad y otra. Consideremos elfirst row X second column, aquí podemos la relación entre pelvic_incidencey pelvic_tilt.
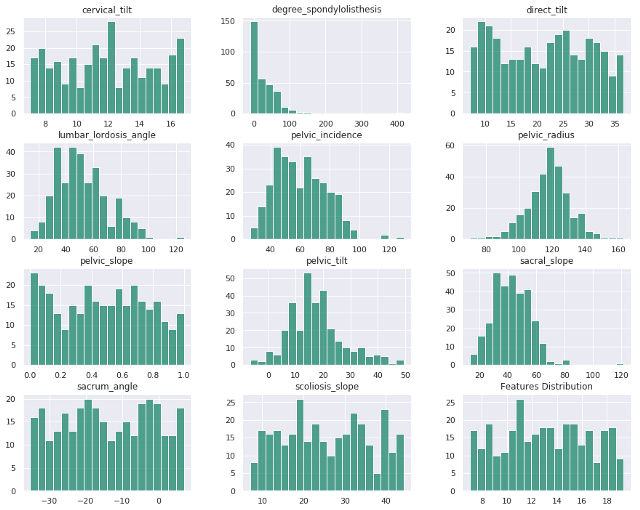
Visualizar características con histograma:
A Histograma es el gráfico más comúnmente utilizado para mostrar las distribuciones de frecuencia.
dataset.hist(figsize=(15,12),bins = 20, color="#007959AA")
plt.title("Features Distribution")
plt.show()

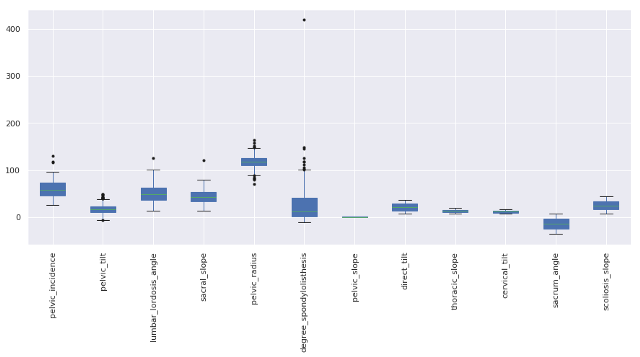
Detección y eliminación de Outliers
plt.subplots(figsize=(15,6))
dataset.boxplot(patch_artist=True, sym=”k.”)
plt.xticks(rotation=90)
Eliminando Outliers:
# we use tukey method to remove outliers.
# whiskers are set at 1.5 times Interquartile Range (IQR)
def remove_outlier(feature):
first_q = np.percentile(X[feature], 25)
third_q = np.percentile(X[feature], 75)
IQR = third_q - first_q
IQR *= 1.5
minimum = first_q - IQR # the acceptable minimum value
maximum = third_q + IQR # the acceptable maximum value
mean = X[feature].mean()
"""
# any value beyond the acceptance range are considered
as outliers.
# we replace the outliers with the mean value of that
feature.
"""
X.loc[X[feature] < minimum, feature] = mean
X.loc[X[feature] > maximum, feature] = mean
# taking all the columns except the last one
# last column is the label
X = dataset.iloc[:, :-1]
for i in range(len(X.columns)):
remove_outlier(X.columns[i])
Después de eliminar outliers:

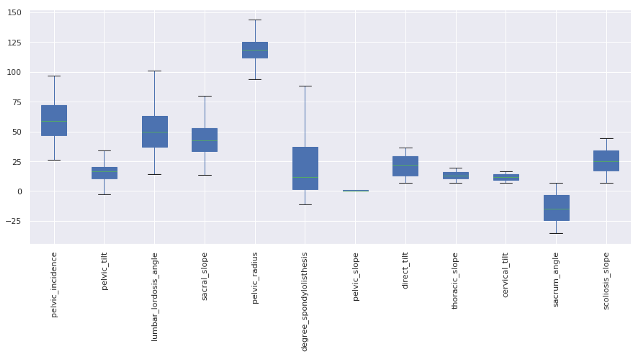
Escalado de features o características:
El escalado de features a través de la estandarización (o normalización Z-score) puede ser un paso de preprocesamiento importante para muchos algoritmos de aprendizaje automático.
Nuestro dataset contiene características que varían mucho en magnitudes, unidades y rango.
Pero dado que la mayoría de los algoritmos de aprendizaje automático utilizan la distancia euclidiana entre dos puntos de datos en sus cálculos, esto creará un problema.
Para evitar este efecto, es necesario que todas las características se encuentren en el mismo nivel de magnitud. Esto se puede lograr mediante escalado de features .
scaler = MinMaxScaler()
scaled_data = scaler.fit_transform(X)
scaled_df = pd.DataFrame(data = scaled_data, columns = X.columns)
scaled_df.head()
Para ver el dataset completo ingrese aquí.
Label Encoding:
Ciertos algoritmos como XGBoost solo pueden tener valores numéricos como sus variables de predicción. Por lo tanto, necesitamos codificar nuestros valores categóricos.
LabelEncoder del paquete sklearn.preprocessing codifica etiquetas con valores entre 0 y n_classes-1.
label = dataset["class"]
encoder = LabelEncoder()
label = encoder.fit_transform(label)
Entrenamiento y evaluación del modelo:
X = scaled_df
y = label
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.15, random_state=0)
clf_gnb = GaussianNB()
pred_gnb = clf_gnb.fit(X_train, y_train).predict(X_test)
accuracy_score(pred_gnb, y_test)
# Out []: 0.8085106382978723
clf_svc = SVC(kernel="linear") pred_svc = clf_svc.fit(X_train, y_train).predict(X_test) accuracy_score(pred_svc, y_test)
# Out []: 0.7872340425531915
clf_xgb = XGBClassifier() pred_xgb = clf_xgb.fit(X_train, y_train).predict(X_test) accuracy_score(pred_xgb, y_test)
# Out []: 0.8297872340425532
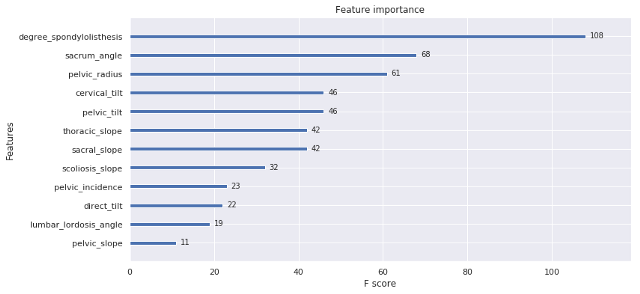
Importancia de Feature:
fig, ax = plt.subplots(figsize=(12, 6))
plot_importance(clf_xgb, ax=ax)

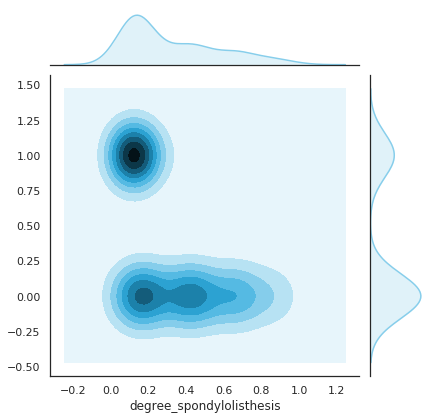
Marginal Plot
Marginal Plot nos permite estudiar la relación entre 2 variables numéricas. El gráfico central muestra su correlación.
Permite visualizar la relación entre degree_spondylolisthesis y class:
sns.set(style="white", color_codes=True)
sns.jointplot(x=X["degree_spondylolisthesis"], y=label, kind='kde', color="skyblue")

Gracias por leer. 🙂
Para obtener el código completo, visite Kaggle o Google Colab .